Qué herramientas permiten la predicción de proteínas
La predicción de proteínas es una de las áreas más fascinantes y complejas de la biología molecular, donde los investigadores buscan desentrañar los misterios de cómo las secuencias de aminoácidos se pliegan para formar estructuras funcionales en los organismos vivos. Este proceso es crucial para entender el funcionamiento de las células, y su relevancia se extiende a campos como la biomedicina, la biotecnología y la farmacología. La capacidad de predecir la estructura y función de las proteínas a partir de su secuencia de aminoácidos puede revolucionar el desarrollo de terapias y tratamientos personalizados.
En este artículo, exploraremos las diversas herramientas y enfoques que han sido desarrollados para la predicción de proteínas. Desde métodos tradicionales basados en la comparación de secuencias hasta técnicas avanzadas que emplean inteligencia artificial y aprendizaje automático, cada uno de estos métodos ha contribuido a avanzar en nuestro entendimiento de cómo las proteínas funcionan. Analizaremos cómo estas herramientas están cambiando la forma en que los científicos abordan la investigación en este ámbito y cuáles son sus aplicaciones prácticas en la actualidad.
- Introducción a la predicción de proteínas
- Métodos tradicionales de predicción de proteínas
- La llegada de la inteligencia artificial y el aprendizaje automático
- Otras herramientas importantes en la predicción de proteínas
- Aplicaciones prácticas en la investigación y medicina
- Retos y futuro de la predicción de proteínas
- Conclusión
Introducción a la predicción de proteínas
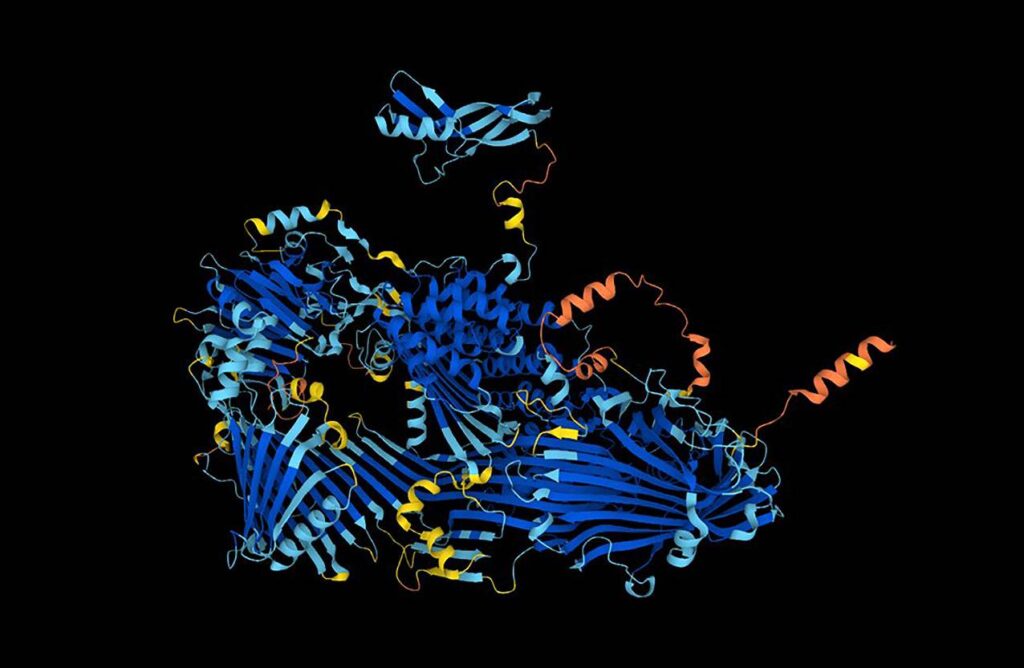
La predicción de proteínas se refiere a la identificación de la estructura tridimensional que adoptará una cadena de aminoácidos determinada. Esta actividad es fundamental porque la función de una proteína está estrechamente relacionada con su estructura. Comprender la relación entre la secuencia de aminoácidos y la estructura que esta adoptará es un objetivo central en la biología molecular, y ha llevado al desarrollo de diversas metodologías. Predecir cómo una proteína se pliega en un espacio tridimensional ayudaría enormemente en la investigación biomédica y en el diseño de nuevas moléculas terapéuticas.
Desde la secuenciación del ADN y las proteínas, los científicos han tenido acceso a un vasto conjunto de datos que les permite entender mejor cómo se relacionan las secuencias y las estructuras. A medida que la cantidad de información disponible ha crecido, también lo han hecho las técnicas y herramientas para su análisis. Las herramientas de predicción de proteínas incluyen desde programas básicos de alineación de secuencias hasta complicadas simulaciones de dinámica molecular que utilizan algoritmos avanzados. En la actualidad, la combinación de estas herramientas ofrece una visión más completa y precisa de la predicción proteica, esencial para múltiples aplicaciones, desde la investigación básica hasta la práctica clínica.
Métodos tradicionales de predicción de proteínas
Los métodos tradicionales para la predicción de proteínas se basan en la comparación de secuencias y la estadística. Uno de los enfoques más comunes es el uso de **homología**. Este método se basa en la premisa de que las proteínas que comparten similitudes en sus secuencias probablemente también compartirán estructuras y funciones. Programas como **BLAST** (Basic Local Alignment Search Tool) permiten a los investigadores encontrar regiones similares dentro de las secuencias proteicas y, en muchos casos, inferir la estructura a partir de proteínas ya conocidas.
Otro método tradicional involucra la utilizacion de **modelos de homología**. Estos modelos se construyen al tomar la estructura de una proteína conocida, conocida como plantilla, y alinear la secuencia de la proteína de interés a esta plantilla. Gracias a este proceso, los investigadores pueden predecir la estructura tridimensional de la proteína en cuestión. Herramientas como **MODELLER** y **SWISS-MODEL** son ejemplos de software que permiten generar estos modelos de homología.
A pesar de su utilidad, los métodos tradicionales tienen limitaciones. En ocasiones, encontrar una plantilla adecuada para la homología puede ser complicado si la proteína de interés es muy diferente de las conocidas. Además, la precisión de estos métodos puede variar, especialmente cuando se trata de proteínas complejas con múltiples dominios o aquellas que cambia de forma en distintas condiciones.
La llegada de la inteligencia artificial y el aprendizaje automático
En los últimos años, hemos visto un auge en la aplicación de la inteligencia artificial (IA) y el aprendizaje automático en el campo de la predicción de proteínas. Estas tecnologías han permitido abordar algunas de las limitaciones de los métodos tradicionales y han mejorado la precisión de las predicciones. Uno de los innovadores enfoques es el uso de redes neuronales profundas, que han demostrado ser capaces de aprender patrones complejos a partir de grandes volúmenes de datos.
Una herramienta de predicción que ha capturado la atención mundial es **AlphaFold**, desarrollada por DeepMind. Este sistema utiliza un modelo de aprendizaje profundo que ha logrado realizar predicciones de estructuras de proteínas con una precisión sin precedentes. Las capacidades de AlphaFold han sido confirmadas en la competición **CASP** (Critical Assessment of protein Structure Prediction), donde demostró que puede predecir estructuras tridimensionales de proteínas con un alto nivel de precisión solo a partir de su secuencia de aminoácidos.
El impacto de AlphaFold ha sido significativo, no solo en el ámbito académico, sino que también ha abierto nuevas oportunidades en la industria farmacéutica y bioquímica. Con esta herramienta, los investigadores pueden predecir estructuras de proteínas que antes eran difíciles de resolver experimentalmente y acelerar el proceso de descubrimiento de medicamentos.
Otras herramientas importantes en la predicción de proteínas
Aparte de AlphaFold, existen otras herramientas que también han sido desarrolladas para la predicción de proteínas. Algunas de ellas se centran en diferentes aspectos del plegamiento y la función de las proteínas. Por ejemplo, **Rosetta** es un conjunto de programas multipropósito que se utiliza para predecir y diseñar estructuras de proteínas. Implica la simulación de varios escenarios de plegamiento y permite a los investigadores explorar diferentes conformaciones posibles antes de dejarse guiar por el método experimental.
Otras herramientas, como **PSSpred** y **NetSurfP**, se enfocan en la predicción de características específicas de la estructura proteica, como la localización en la membrana, la estructura secundaria y más. Estas herramientas son importantes ya que proporcionan información adicional que puede ser valiosa durante el proceso de diseño y análisis de proteínas.
Aplicaciones prácticas en la investigación y medicina
La aplicación de herramientas de predicción de proteínas va más allá del mero interés académico; también juega un papel vital en el avance de la medicina personalizada y la biotecnología. En la investigación médica, la capacidad de prever múltiples **estructuras** de proteínas puede ayudar a identificar nuevos **marcadores** de enfermedades e **inmunógenos** que pueden ser utilizados en la vacunación.
En el desarrollo de fármacos, la predicción de la estructura de proteínas diana permite a los científicos diseñar inhibidores que pueden unirse de manera efectiva a estas proteínas, bloqueando su actividad y potencialmente deteniendo el avance de enfermedades. Así, instrumentos como **ZINC** y **AutoDock** permiten analizar interacciones y facilitar el descubrimiento de nuevos agentes terapéuticos desde un enfoque computacional.
Además, estas herramientas son esenciales para comprender cómo las mutaciones en las proteínas pueden conducir a enfermedades. Al emplear predicciones de estructura, los investigadores pueden modelar cómo podría cambiar la función de una proteína en respuesta a alteraciones genéticas, brindando así información vital sobre la patogenia de diversas enfermedades, incluidas muchas formas de cáncer y trastornos genéticos.
Retos y futuro de la predicción de proteínas
A pesar de los avances significativos, la predicción de proteínas aún enfrenta varios desafíos. Uno de los principales retos es la **complejidad del plegamiento proteico**. Mientras que las herramientas actuales pueden predecir estructuras en muchos casos, el plegamiento espontáneo en sí mismo es un proceso extremadamente complejo que todavía no se comprende del todo. A medida que las proteínas interaccionan con otras moléculas, su estructura puede cambiar, lo que añade un nivel de dificultad a las predicciones.
Asimismo, la calidad y cantidad de datos de calidad también son aspectos que limitan la evolución de la predicción de proteínas. Si bien el acceso a grandes bancos de datos ha mejorado, todavía hay un vacío en ciertas áreas, lo que puede afectar la capacitación de los modelos de aprendizaje automático. Por lo tanto, el futuro de la predicción de proteínas dependerá de la combinación de avances tecnológicos y la generación de más datos de alta calidad, facilitando así el entrenamiento de modelos más robustos y precisos.
Conclusión
La predicción de proteínas ha avanzado a pasos agigantados gracias a la introducción de herramientas más sofisticadas que van desde métodos tradicionales hastaenciales en IA y aprendizaje automático. Estas innovaciones han alterado el panorama de la biología molecular, permitiendo a los científicos explorar el mundo de las proteínas de formas que antes eran inalcanzables. Las aplicaciones prácticas en medicina y biotecnología demuestran el potencial vasto de estas herramientas.No obstante, el campo sigue enfrentando retos significativos que deberán ser abordados para seguir avanzando en la predicción y comprensión del comportamiento de las proteínas.
Con la continua evolución de la tecnología y la colaboración entre diversos campos científicos, es de esperar que el futuro de la predicción de proteínas se presenten con nuevas oportunidades y descubrimientos que mejoren nuestra comprensión del mundo biológico y contribuyan a la mejora de la salud humana.
Si quieres conocer otros artículos parecidos a Qué herramientas permiten la predicción de proteínas puedes visitar la categoría Herramientas.




Deja una respuesta