
Algoritmos destacados en la historia de la bioinformática
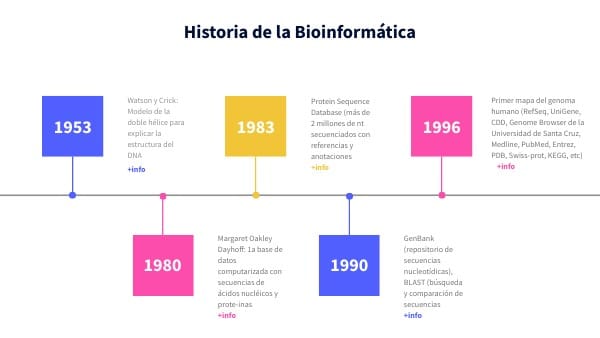
La bioinformática ha evolucionado de manera asombrosa en las últimas décadas, transformándose en una disciplina fundamental para el avance de la biología molecular y la genética. Esta fusión de biología, matemáticas, informática y estadística ha dado lugar a la creación de herramientas y algoritmos que permiten analizar grandes volúmenes de datos biológicos. El impacto de estos algoritmos ha sido significativo, no solo en la investigación académica, sino también en aplicaciones industriales y clínicas, como la medicina personalizada y el descubrimiento de fármacos.
Este artículo explorará algunos de los algoritmos más destacados en la historia de la bioinformática, analizando su desarrollo, importancia y aplicaciones. Desde la secuenciación del ADN hasta la predicción de estructuras proteicas, cada uno de estos algoritmos ha contribuido a un nuevo entendimiento de la vida a nivel molecular y ha abierto nuevas vías para la investigación biomédica. Así, a lo largo del texto, examinaremos no solo su funcionalidad, sino también el contexto en el que surgieron y su impacto en el avance de la ciencia.
- El algoritmo de Needleman-Wunsch y la alineación de secuencias
- BLAST: El algoritmo para la búsqueda de similitudes
- Algoritmos de predicción de estructuras proteicas: PDB y AlphaFold
- Las redes neuronales en la predicción y análisis de datos biológicos
- CRISPR y su algoritmo de edición genética
- Conclusión: La trayectoria de los algoritmos en bioinformática
El algoritmo de Needleman-Wunsch y la alineación de secuencias
Introducido en 1970 por Saul B. Needleman y Christian D. Wunsch, el algoritmo de Needleman-Wunsch es uno de los más importantes en bioinformática por su capacidad para realizar alineamientos de secuencias de proteínas y ADN. Este algoritmo utiliza un enfoque de programación dinámica para encontrar la mejor alineación global entre dos secuencias, lo que permite identificar regiones conservadas entre ellas. Su utilización ha sido crucial en estudios evolutivos, donde la comparación de secuencias es fundamental para inferir relaciones filogenéticas.
A lo largo de los años, el algoritmo ha sido optimizado y extendido, apareciendo versiones que pueden manejar múltiples secuencias al mismo tiempo, como es el caso del método de alineación múltiple de secuencias. Sin embargo, la esencia del algoritmo de Needleman-Wunsch radica en su capacidad para introducir un costo por desajuste o por inserciones y deleciones, lo que lo hace invaluable para detectar similitudes que podrían no ser evidentes a simple vista. En el contexto actual, su uso sigue siendo primordial para abordar cuestiones en la evolución y la estructura de proteínas, ofreciendo herramientas para la predicción de funciones biológicas basadas en la similaridad de secuencias.
BLAST: El algoritmo para la búsqueda de similitudes
El algoritmo BLAST (Basic Local Alignment Search Tool) fue desarrollado por Stephen Altschul y sus colegas en 1990 y ha revolucionado la forma en que los científicos buscan homología en bases de datos de secuencias. A diferencia del algoritmo de Needleman-Wunsch, que hace un alineamiento global, BLAST se centra en encontrar alineaciones locales, lo que le permite identificar rápidamente segmentos similares dentro de secuencias más largas. Esto resulta extremadamente útil en estudios de genómica, donde se pueden encontrar patrones conservados en secuencias que, de otro modo, podrían parecer aleatorias.
El funcionamiento de BLAST se basa en la búsqueda de palabras (k-mers), pequeñas secuencias de nucleótidos o aminoácidos, para ampliar la búsqueda de alineaciones en bases de datos. Este enfoque ha permitido que BLAST sea increíblemente rápido y eficiente, lo cual ha sido un factor determinante en su adopción a gran escala en la comunidad científica. Además, BLAST ha facilitado el análisis de secuencias de organismos no secuenciados, proporcionando una forma de predecir la función de genes y proteínas desconocidas al compararlas con secuencias de organismos bien estudiados. La versatilidad de BLAST ha contribuido al surgimiento de bases de datos como GenBank, que recogen miles de millones de secuencias de todo tipo, permitiendo a los investigadores profundizar en la comprensión de la diversidad genética.
Algoritmos de predicción de estructuras proteicas: PDB y AlphaFold
La predicción de estructuras proteicas ha sido otro campo en el que los algoritmos han realizado contribuciones excepcionales. La base de datos Protein Data Bank (PDB) alberga estructuras proteicas conocidas, y el desarrollo de algoritmos para predecir estas estructuras ha sido un objetivo primordial. Tradicionalmente, los métodos de modelado homólogo se utilizaban para inferir la estructura de una proteína en base en la comparación con proteínas cuyas estructuras eran conocidas. Sin embargo, en los últimos años, hemos sido testigos de un avance significativo con la llegada de AlphaFold, un sistema basado en aprendizaje profundo desarrollado por DeepMind.
AlphaFold ha demostrado ser capaz de predecir la estructura de proteínas con una precisión comparable a la que se obtiene mediante cristalografía de rayos X, pero en un tiempo mucho menor. Este avance ha sido verdaderamente revolucionario, ya que permite investigar funciones biológicas y enfermedades a un nivel que antes solo era posible a través de técnicas experimentales complejas y costosas. La capacidad de AlphaFold para realizar predicciones con alta precisión ha puesto de relieve la importancia del aprendizaje automático en bioinformática y ha abierto nuevas puertas en la investigación médica, especialmente en el ámbito del diseño de fármacos y el tratamiento de enfermedades.
Las redes neuronales en la predicción y análisis de datos biológicos
El uso de redes neuronales en el análisis de datos biológicos representa una de las tendencias más emocionantes en bioinformática. Estas técnicas de aprendizaje profundo han demostrado ser efectivas en la clasificación de imágenes, la predicción de estructuras y el análisis de datos clínicos. Las redes neuronales convolucionales, en particular, se están utilizando en el análisis de imágenes de microscopía y en la identificación de patrones en grandes conjuntos de datos genómicos.
Además, las redes neuronales recurrentes están siendo aplicadas con éxito en la predicción de secuencias, donde la naturaleza secuencial de la información biológica se puede modelar de manera eficiente. Estos algoritmos son capaces de aprender representaciones complejas y no lineales de datos, lo que enriquece enormemente nuestra capacidad para identificar relaciones y patrones que podrían no ser evidentes con métodos estadísticos convencionales. La integración de estas tecnologías en la bioinformática promete acelerar la investigación biológica de manera sin precedentes, lo que facilita el descubrimiento de nuevas terapias y la mejora de la salud pública.
CRISPR y su algoritmo de edición genética
Uno de los desarrollos más impactantes en biotecnología y bioinformática ha sido el sistema CRISPR-Cas9, que permite la edición de genes de manera precisa y eficiente. Este sistema se basa en un conjunto de herramientas que utilizan RNA guiado para dirigir una nucleasa a un sitio específico en el ADN, donde puede realizar cortes y, posteriormente, ser reparado de maneras que pueden alterar la función genética. Esto ha abierto un mundo de posibilidades en la manipulación genética, con aplicaciones que van desde la agricultura hasta el tratamiento de enfermedades genéticas en humanos.
El desarrollo de algoritmos que optimizan y predicen el éxito de las ediciones CRISPR se ha convertido en un área de investigación activa, donde se busca maximizar la especificidad y minimizar los efectos no deseados. Técnicas como la modelación computacional se utilizan para prever cómo se comportarán los fragmentos de RNA en el interior de la célula, permitiendo a los investigadores diseñar experimentos más eficientes. El impacto de CRISPR en la bioinformática es evidente, ya que ha ayudado a abrir un camino hacia la personalización de tratamientos médicos y la mejora de cultivos, evidenciando el poder de los algoritmos en la innovación científica.
Conclusión: La trayectoria de los algoritmos en bioinformática
La historia de los algoritmos en bioinformática es un testimonio del ingenio humano y de la búsqueda incesante para comprender la complejidad de la vida a nivel molecular. Desde el algoritmo de Needleman-Wunsch hasta las innovaciones en aprendizaje automático y edición genética con CRISPR, cada uno de estos desarrollos ha contribuido a ampliar nuestras capacidades para analizar e interpretar datos biológicos. A medida que la ciencia avanza y las tecnologías evolucionan, es probable que sigamos viendo nuevas técnicas y mejoras en los algoritmos actuales, lo que sin duda llevará a descubrimientos científicos aún más sorprendentes en el futuro.
La bioinformática, impulsada por estos algoritmos, se ha convertido en una piedra angular en la ciencia contemporánea, y su relevancia solo aumentará a medida que enfrentemos nuevos desafíos en la salud, la biotecnología y la sostenibilidad. A medida que las herramientas se vuelven más sofisticadas y accesibles, el potencial para la innovación en este campo es ilimitado, y los algoritmos seguirán siendo fundamentales en nuestra búsqueda por desentrañar los misterios de la biología.
Si quieres conocer otros artículos parecidos a Algoritmos destacados en la historia de la bioinformática puedes visitar la categoría Algoritmos.


Deja una respuesta