
Cómo usar Galaxy de forma efectiva para análisis científicos
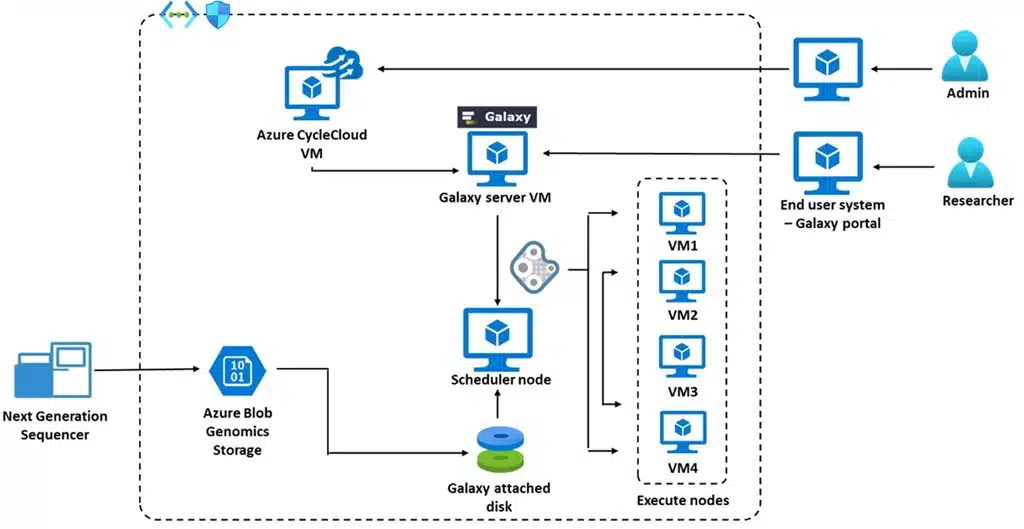
En el mundo de la investigación científica y la bioinformática, el acceso a herramientas que faciliten el análisis de datos es fundamental. Uno de los recursos más valiosos en este ámbito es Galaxy, una plataforma web de código abierto que proporciona un entorno accesible y flexible para la implementación de flujos de trabajo relacionados con el análisis de datos biológicos. Su interfaz amigable y su capacidad para integrar diferentes herramientas han transformado la forma en que los científicos abordan el análisis de datos masivos, permitiendo a los investigadores de diversos niveles técnicos realizar análisis complejos sin la necesidad de programar.
Este artículo tiene como objetivo ofrecer un enfoque detallado sobre cómo utilizar Galaxy de manera efectiva en el ámbito del análisis científico. A lo largo de las siguientes secciones, exploraremos las características principales de Galaxy, su instalación y configuración, así como ejemplos prácticos de flujos de trabajo. Además, se abordará cómo garantizar la reproducibilidad de los análisis y se discutirán los avances futuros en el uso de esta plataforma. Al final, los lectores contarán con un amplio entendimiento sobre cómo maximizar el potencial de Galaxy en su investigación científica.
- Comprendiendo Galaxy: una panorámica del software
- Configuración inicial de Galaxy: instalación y requisitos
- Creación de flujos de trabajo: cómo estructurar el análisis en Galaxy
- Reproducibilidad en el análisis científico: cómo Galaxy aborda este desafío
- Avances futuros y el potencial de Galaxy en la investigación científica
- Conclusión: el futuro de Galaxy en el análisis científico
Comprendiendo Galaxy: una panorámica del software
Galaxy es un sistema de software que permite a los científicos gestionar, analizar e interpretar datos a través de una interfaz gráfica de usuario (GUI). Al contrario de otros sistemas que requieren un amplio conocimiento en programación, Galaxy está diseñado para ser accesible a los biólogos y otros científicos que pueden no tener formación en informática. Esto es especialmente relevante en el contexto actual, donde los datos generados por tecnologías como la secuenciación de próxima generación (NGS) son enormes y complejos.
En su núcleo, Galaxy permite la creación de flujos de trabajo personalizables mediante el uso de herramientas que pueden ser agregadas y encadenadas a través de una interfaz intuitiva. Este entorno disminuye las barreras de entrada para el análisis bioinformático, permitiendo a cualquier persona con conocimientos básicos en biología realizar análisis sofisticados. Además, Galaxy soporta una amplia variedad de formatos de datos y herramientas, lo que lo convierte en una solución versátil para diferentes áreas de investigación.
Configuración inicial de Galaxy: instalación y requisitos
Antes de comenzar a utilizar Galaxy, es fundamental entender los requisitos necesarios para su instalación. Existen diferentes maneras de instalar Galaxy, ya sea en un servidor local, en un clúster de computación o utilizando la versión basada en la nube. La elección del método dependerá de la infraestructura disponible y de las preferencias del usuario.
Para instalar Galaxy localmente, se requiere de un sistema operativo compatible, preferentemente Linux o macOS. Además, es importante contar con software adicional como Python, un servidor web y una base de datos, típicamente PostgreSQL. Los usuarios también pueden optar por utilizar contenedores Docker, que simplifican el proceso de instalación al permitir ejecutar aplicaciones en entornos aislados. Esta es una opción recomendada para aquellos que están comenzando y desean evitar complicaciones técnicas.
Una vez que se ha completado la instalación, es crucial configurar adecuadamente la base de datos y los diversos parámetros de entorno para optimizar el rendimiento. La correcta configuración del entorno no solo mejora la funcionalidad de Galaxy, sino que también asegura una experiencia de usuario fluida.
Creación de flujos de trabajo: cómo estructurar el análisis en Galaxy
Uno de los aspectos más poderosos de Galaxy es la capacidad de crear flujos de trabajo personalizados que encapsulan todos los pasos necesarios para un análisis específico. Este enfoque modular permite a los científicos implementar múltiples herramientas en un solo flujo de trabajo, facilitando el análisis y la visualización de datos.
Para crear un flujo de trabajo en Galaxy, los usuarios deben seleccionar las herramientas que desean utilizar de la caja de herramientas de Galaxy. La plataforma ofrece una interfaz que permite arrastrar y soltar estas herramientas en el área de trabajo. Cada herramienta puede configurarse con parámetros específicos, como archivos de entrada y opciones de ejecución. Este enfoque visual no solo hace que el proceso sea más intuitivo, sino que también permite una fácil modificación y reutilización de flujos de trabajo existentes.
Una vez que se ha configurado un flujo de trabajo, los usuarios pueden ejecutar todo el análisis de una sola vez, lo que garantiza que todos los pasos se ejecuten en el orden correcto. Además, Galaxy permite la visualización de resultados de cada paso del flujo de trabajo, lo que ayuda a los investigadores a identificar rápidamente posibles problemas o anomalías en el análisis.
Reproducibilidad en el análisis científico: cómo Galaxy aborda este desafío
La reproducibilidad es un principio fundamental en la investigación científica, ya que garantiza que los resultados sean confiables y verificables. Gracias a su enfoque modular y basado en flujos de trabajo, Galaxy facilita la reproducibilidad de los análisis. Cada flujo de trabajo puede ser guardado y compartido con otros usuarios, quienes pueden ejecutar el mismo análisis con sus propios datos utilizando la misma configuración y herramientas.
Además, Galaxy permite la exportación de flujos de trabajo como archivos JSON, que pueden ser almacenados y versionados. Esto significa que los investigadores pueden llevar un registro de las versiones de sus análisis, lo que es particularmente valioso en un entorno en rápida evolución como la investigación biológica. También es posible compartir flujos de trabajo a través de plataformas de colaboración, lo que fomenta el intercambio de conocimiento y metodologías entre científicos.
Avances futuros y el potencial de Galaxy en la investigación científica
A medida que la bioinformática sigue evolucionando, el futuro de Galaxy parece brillante. La comunidad de usuarios y desarrolladores está constantemente trabajando para aumentar la cantidad de herramientas disponibles y mejorar la interfaz de usuario. Una de las áreas más prometedoras es la integración de Galaxy con plataformas de aprendizaje automático e inteligencia artificial, lo que podría revolucionar la forma en que se analizan y procesan grandes volúmenes de datos.
Además, se prevé que las futuras versiones de Galaxy incorporen mayor soporte para tecnologías emergentes, como el análisis de datos de genómica única y estudios multi-ómicos. Esto ampliará aún más el alcance y la utilidad de Galaxy en diversas disciplinas científicas. Asimismo, el enfoque colaborativo en la plataforma puede fomentar la formación de comunidades de investigación en torno a temas específicos, lo que puede potenciar avances significativos en el campo.
Conclusión: el futuro de Galaxy en el análisis científico
Galaxy se establece como una herramienta revolucionaria en el análisis científico y la bioinformática. Su accesibilidad, flexibilidad y soporte para flujos de trabajo personalizados permiten a los investigadores abordar el análisis de datos complejos de manera eficaz y reproducible. A medida que la comunidad de usuarios y desarrolladores continúa creciendo, se espera que Galaxy se implemente en un rango aún más amplio de aplicaciones y disciplinas.
Al fomentar la reproducibilidad y la transparencia en el análisis, Galaxy no solo beneficia a los investigadores individuales, sino que también contribuye a la integridad y confiabilidad de la investigación científica en general. Con la evolución continua de la plataforma, se posiciona como un recurso indispensable para cualquier científico que busque realizar análisis de datos biológicos de manera efectiva y rigurosa.
Si quieres conocer otros artículos parecidos a Cómo usar Galaxy de forma efectiva para análisis científicos puedes visitar la categoría Software.




Deja una respuesta