Crear un flujo de trabajo reproducible en bioinformática
La bioinformática se ha convertido en un pilar fundamental en el ámbito de la investigación biológica, fusionando el poder de la computación y la biología para desentrañar los secretos del ADN, las proteínas y las interacciones celulares. Con la creciente cantidad de datos biológicos generados por tecnologías como la secuenciación de nueva generación, la necesidad de establecer flujos de trabajo eficientes y reproducibles se ha vuelto crítica. Un flujo de trabajo bien diseñado no solo ahorra tiempo, sino que también mejora la calidad de los resultados obtenidos, permitiendo a los investigadores replicar experimentos y validar hallazgos con mayor facilidad.
En este artículo, vamos a explorar cómo crear un flujo de trabajo reproducible en bioinformática, enfocándonos en las mejores prácticas y herramientas que pueden ser utilizadas para mejorar la reproducibilidad de los análisis. A medida que profundicemos en este tema, discutiremos diversos aspectos, desde la elección de herramientas y lenguajes de programación hasta la documentación adecuada y la gestión de datos. Preparémonos para un viaje por las principales consideraciones y estrategias que, sin duda, marcarán la diferencia en la calidad de los trabajos realizados en el apasionante campo de la bioinformática.
- La importancia de la reproducibilidad en la bioinformática
- Herramientas y lenguajes para la creación de flujos de trabajo reproducibles
- Documentación y metadatos: la clave de la claridad
- Gestión de datos: almacenamiento y organización
- Pruebas unitarias y validación de análisis
- Conclusión: el futuro de la bioinformática y la reproducibilidad
La importancia de la reproducibilidad en la bioinformática
La reproducibilidad se refiere a la capacidad de un investigador para obtener los mismos resultados al repetir un experimento o análisis utilizando los mismos métodos y datos. En bioinformática, esto es especialmente crucial debido a la complejidad de los datos biológicos y la variedad de enfoques analíticos disponibles. La falta de reproducibilidad puede dar lugar a resultados erróneos, la pérdida de confianza en los hallazgos y, en el peor de los casos, la promulgación de conclusiones equivocadas que pueden afectar futuras investigaciones y aplicaciones en medicina, genética y otras áreas.
Establecer un flujo de trabajo reproducible implica documentar cada paso del proceso de análisis, desde la adquisición de datos hasta la publicación de resultados. Esto incluye no solo la elección de los métodos y herramientas, sino también la versión de los software utilizados, las configuraciones específicas y el código encargado de realizar los análisis. Al seguir estas prácticas, los investigadores no solo pueden facilitar la revisión por pares, sino que también contribuyen a la creación de ciencia abierta y accesible, donde el conocimiento es compartido y construido colectivamente.
Herramientas y lenguajes para la creación de flujos de trabajo reproducibles
Existen numerosas herramientas y lenguajes que pueden facilitar el desarrollo de un flujo de trabajo reproducible. Por un lado, lenguajes como R y Python son ampliamente utilizados en bioinformática debido a su versatilidad y la disponibilidad de paquetes específicos que simplifican el análisis de datos biológicos. Con bibliotecas como Bioconductor en R y Biopython en Python, los investigadores pueden realizar análisis complejos con relativamente poco esfuerzo, lo que reduce la probabilidad de errores y mejora la reproducibilidad.
Además de los lenguajes de programación, el uso de sistemas de gestión de versiones como Git se ha vuelto imprescindible. Git permite realizar un seguimiento de los cambios en el código y en los documentos asociados, facilitando la colaboración con otros investigadores y permitiendo regresar a versiones anteriores en caso de que seas necesario. Los repositorios en plataformas como GitHub no solo almacenan el código, sino que también permiten a otros acceder al trabajo, reproducir los análisis y, en última instancia, validar los resultados.
Documentación y metadatos: la clave de la claridad
La documentación es un componente esencial de cualquier flujo de trabajo reproducible. Cada etapa del análisis debe estar acompañada por una descripción clara y concisa que explique el propósito de esa etapa, los datos utilizados y las herramientas empleadas. Esto se puede hacer a través de un archivo README que esté incluido en el repositorio del proyecto. Además, incorporar metadatos sobre los datos biológicos analizados proporciona un contexto que puede ser vital para otros investigadores que deseen reproducir el trabajo.
Utilizar herramientas de documentación como Markdown o Sphinx permite presentar la información de manera estructurada y fácil de seguir. Es vital indicar las versiones de los paquetes y bibliotecas utilizadas, así como cualquier configuración particular que haya influido en los resultados finales. Implementar prácticas de documentación como esta no solo es beneficioso para otros, sino que también ayuda a los propios investigadores a recordar metodologías al pasar el tiempo.
Gestión de datos: almacenamiento y organización
La gestión de datos es un aspecto fundamental en la bioinformática, ya que el análisis depende enormemente de la calidad y organización de los datos utilizados. El primer paso en la gestión de datos es establecer un esquema de nomenclatura coherente y fácil de seguir. Esto asegura que los archivos y carpetas sean fácilmente identificables y accesibles para cualquier persona que intente reproducir el análisis.
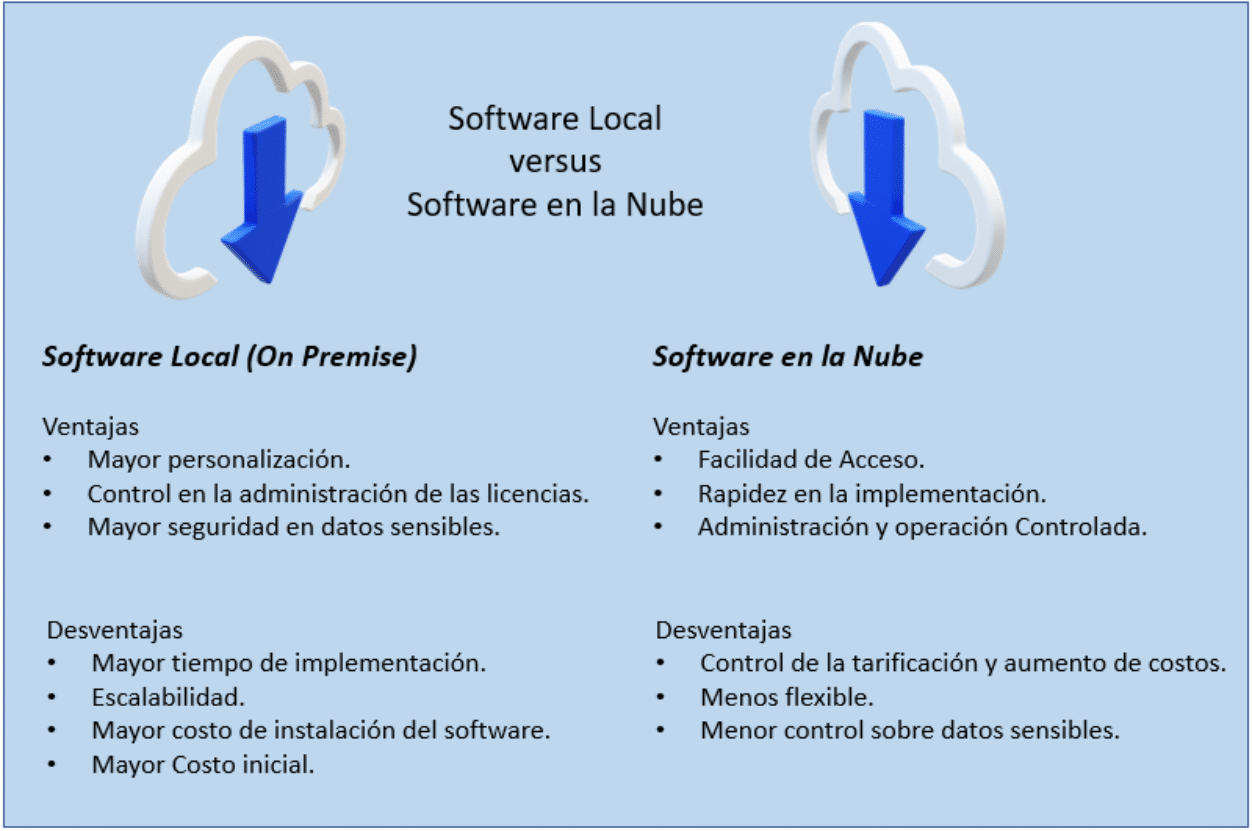
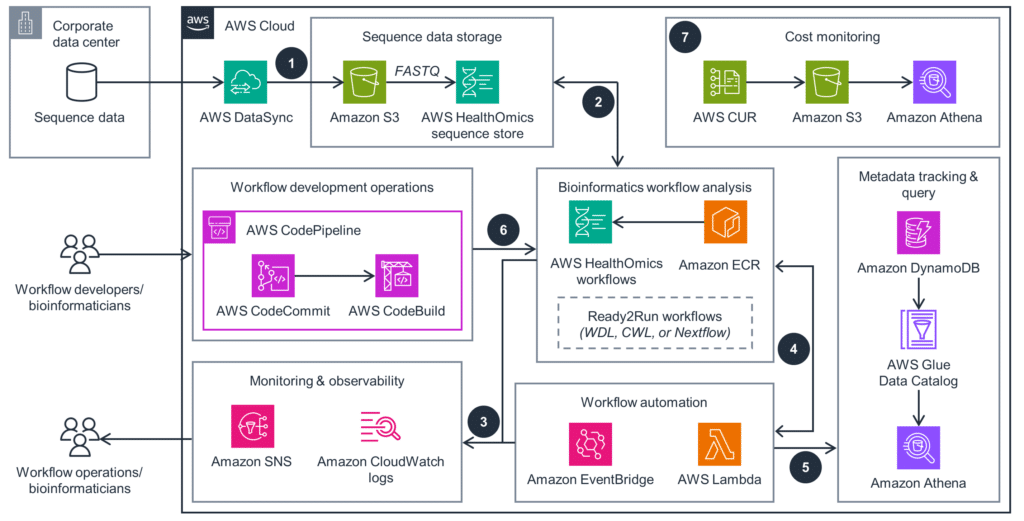
Además, es esencial emplear plataformas de almacenamiento que garanticen la seguridad y accesibilidad de los datos. Los servicios en la nube, como AWS o Google Cloud, ofrecen opciones escalables para almacenar grandes volúmenes de información, además de contar con herramientas que permiten un fácil acceso y manipulación de estos datos. Adicionalmente, siempre es recomendable mantener copias de seguridad de los datos originales, ya que pueden ser necesarios para validar o repetir ciertos pasos del análisis en el futuro.
Pruebas unitarias y validación de análisis
Las pruebas unitarias se utilizan para verificar que cada componente del flujo de trabajo se desempeñe según lo previsto. Esto es especialmente relevante en bioinformática, donde un pequeño error en el código puede llevar a resultados completamente erróneos. Implementar un marco de pruebas, como pytest en Python, permite a los investigadores garantizar que su código funcione como se espera y que los cambios realizados en el mismo no generen efectos negativos en el flujo de trabajo general.
Aparte de las pruebas unitarias, es fundamental también el proceso de validación de análisis. Esto implica replicar los análisis en conjuntos de datos independientes o comparar resultados con aquellos obtenidos por otros métodos establecidos. La validación no solo proporciona una verificación adicional de la reproducibilidad, sino que también fortalece la credibilidad del trabajo presentado en publicaciones científicas.
Conclusión: el futuro de la bioinformática y la reproducibilidad
Crear un flujo de trabajo reproducible en bioinformática no es solo una práctica recomendada, sino una necesidad creciente en un campo que avanza rápidamente. La reproducibilidad asegura que los hallazgos no sean solo una ilusión, sino resultados válidos que pueden ser utilizados y construidos a partir de ellos en otras investigaciones. A través de la adopción de herramientas adecuadas, documentación minuciosa, gestión de datos efectiva y la implementación de pruebas unitarias, los bioinformáticos pueden contribuir a un ecosistema más abierto y colaborativo.
La integración de estas prácticas mejorará no solo la calidad de los análisis realizados, sino que también impulsará la confianza entre la comunidad científica y el público en general. Frente a desafíos como la cantidad creciente de datos y la complejidad de los análisis involucrados, la reproducibilidad se presenta como un faro que puede guiar la investigación bioinformática hacia un futuro más sólido y confiable. A medida que avanzamos en la exploración del vasto mapa de la biología, asegurémonos de que nuestros caminos sean claros y accesibles para todos los que deseen unirse a este prometedor viaje.
Si quieres conocer otros artículos parecidos a Crear un flujo de trabajo reproducible en bioinformática puedes visitar la categoría Software.




Deja una respuesta