Definición y Función de un Pipeline en Bioinformática
La bioinformática se ha convertido en un campo fundamental para la investigación biológica, ya que permite el análisis y la interpretación de enormes volúmenes de datos biológicos. En este contexto, la utilización de pipelines se ha vuelto esencial para la gestión eficiente de estos datos, facilitando la automatización de procesos que en el pasado requerían un esfuerzo significativamente mayor. Pero, ¿qué es exactamente un pipeline y cuál es su función en bioinformática? A través de este artículo, recorreremos en detalle la definición, la estructura y la importancia de los pipelines en el análisis de datos biológicos.
Este artículo está diseñado para ofrecerte una comprensión profunda sobre el concepto de pipeline en bioinformática. Veremos no solo su definición, sino también cómo se implementan en la práctica y las ventajas que proporcionan al manejo de datos. A lo largo de secciones bien estructuradas, exploraremos los distintos componentes que conforman un pipeline, así como ejemplos de su aplicación en el análisis de datos genómicos, transcriptómicos y proteómicos. La meta es proporcionar un recurso completo que te permita entender y aplicar estos conocimientos en tus proyectos de investigación.
¿Qué es un Pipeline en Bioinformática?
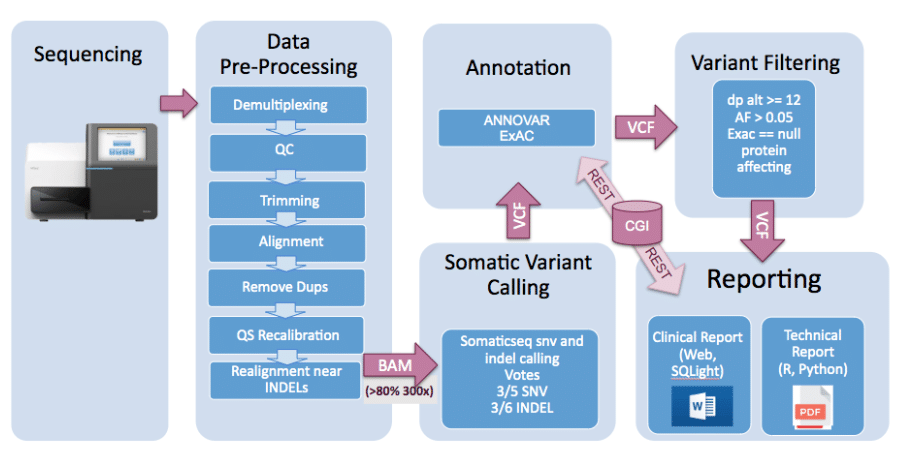
Un pipeline en bioinformática es una secuencia de herramientas y procesos que transforma, analiza y organiza datos biológicos de manera automática. Este término se utiliza comúnmente para describir una serie de pasos que se suelen ejecutar de forma consecutiva, donde la salida de un paso se convierte en la entrada del siguiente. El uso de pipelines es crucial para manejar los múltiples flujos de datos generados por tecnologías de secuenciación de última generación y otros métodos de análisis biológicos.
Los pipelines pueden ser simples o complejos, dependiendo del tipo de análisis que se desea llevar a cabo. Por ejemplo, en el análisis de secuencias de ADN, un pipeline típico podría incluir la calidad del control de las secuencias, el alineamiento de las secuencias contra un genoma de referencia, la detección de variantes y, finalmente, la anotación de estas variantes para su interpretación funcional. Cada etapa del pipeline está diseñada para realizar tareas específicas, lo que asegura que cada paso se ejecute de manera eficiente y coherente.
Estructura de un Pipeline: Componentes Clave
La estructura de un pipeline en bioinformática normalmente incluye varios componentes clave. Estos elementos son cruciales para asegurar que el flujo de datos se maneje de manera fluida y que el análisis final sea de alta calidad. Entre los componentes más destacados se encuentran:
1. **Preprocesamiento de Datos**: Esta es la primera etapa en la que se realizan operaciones iniciales, como el control de calidad y el filtrado de datos. En estudios de secuenciación, por ejemplo, es fundamental eliminar secuencias de baja calidad o contaminadas antes de continuar con el análisis.
2. **Alineamiento**: Una vez que los datos han sido preprocesados, el siguiente paso suele ser el alineamiento de las secuencias obtenidas con un genoma de referencia. Este proceso permite identificar la ubicación de las secuencias dentro del genoma y facilitar el análisis de variantes.
3. **Análisis de Variantes**: Este componente se ocupa de la detección de diferencias en la secuencia de nucleótidos entre las muestras analizadas y el genoma de referencia. Es una parte fundamental para entender las variantes genéticas asociadas a enfermedades o características específicas.
4. **Anotación**: Finalmente, el pipeline proporciona información sobre la funcionalidad de las variantes detectadas. Este paso a menudo incluye la comparación de las variantes con bases de datos existentes que catalogan el conocimiento previo sobre genes y su función.
Tipos de Pipelines en Bioinformática
Los pipelines en bioinformática se pueden clasificar en varias categorías, según el tipo de análisis que requieren. A continuación, mencionaremos algunos de los tipos más comunes:
1. **Pipelines de Secuenciación**: Estos son utilizados para procesar datos generados por plataformas de secuenciación, como Illumina o PacBio. Incluyen pasos como la limpieza de datos, alineación y análisis de variantes.
2. **Pipelines de RNA-Seq**: En estudios que implican análisis de ARN, se utilizan pipelines específicos para procesar datos de RNA-Seq. Estos se ocupan de la cuantificación de la expresión génica y la detección de isoformas.
3. **Pipelines de Proteómica**: Este tipo de pipelines se diseñan para analizar datos obtenidos de técnicas de espectrometría de masas. Incluyen pasos de identificación y cuantificación de proteínas, así como la caracterización de interacciones proteína-proteína.
4. **Pipelines de Metagenómica**: Estos facilitan el análisis de datos de comunidades microbianas mediante la secuenciación de material genético extraído de muestras ambientales. Su objetivo es identificar y caracterizar las especies presentes en un entorno específico.
Ventajas de Utilizar Pipelines en Bioinformática
El uso de pipelines presenta varias ventajas en el ámbito de la bioinformática, entre las que destacan:
1. **Automatización**: Los pipelines permiten automatizar procesos que de otro modo serían manuales y propensos a errores. Al ejecutar las herramientas en un orden establecido, se minimiza el riesgo de errores humanos y se incrementa la reproducibilidad del análisis.
2. **Eficiencia**: Al estructurar el análisis en etapas, se puede hacer un uso más eficiente de los recursos computacionales. Esto permite acelerar los tiempos de procesamiento y, en consecuencia, obtener resultados más rápidos.
3. **Reproducibilidad**: Al definir claramente cada etapa del proceso, se facilita la reproducibilidad de los resultados, lo que es esencial en la investigación científica actual. Otros investigadores pueden replicar los análisis siguiendo el mismo pipeline, lo cual es crítico para validar hallazgos.
4. **Facilidad de Uso**: Muchos pipelines están diseñados con interfaces de usuario amigables y documentaciones claras, lo que permite que incluso los investigadores sin experiencia en programación puedan utilizarlos efectivamente.
Ejemplos de Herramientas de Pipelines en Bioinformática
Existen diversas herramientas y plataformas que permiten la creación y ejecución de pipelines en bioinformática. Algunas de las más populares incluyen:
1. **Galaxy**: Esta es una plataforma web que facilita el análisis de datos biológicos a través de la creación de pipelines visuales. Los usuarios pueden combinar diferentes herramientas de análisis sin necesidad de escribir código, lo que la hace accesible a un público más amplio.
2. **Nextflow**: Esta es una herramienta diseñada para crear y ejecutar pipelines reproducibles, que se pueden ejecutar en diversas plataformas de computación, desde el escritorio hasta clústeres de alto rendimiento. Ofrece flexibilidad en la integración de diferentes lenguajes de programación y herramientas.
3. **Snakemake**: Similar a Nextflow, Snakemake ofrece un enfoque centrado en la creación de pipelines basados en scripts de Python. Permite gestionar dependencias entre las distintas etapas del pipeline y garantiza la ejecución eficiente de los procesos definidos.
4. **Cromwell**: Esta es una herramienta de ejecución de workflows que es parte del proyecto Broad Institute. Cromwell permite a los investigadores reproducir análisis complejos utilizando lenguajes de script como WDL (Workflow Description Language).
Consideraciones Finales sobre los Pipelines en Bioinformática
La utilización de pipelines en bioinformática ha cambiado radicalmente la forma en que los datos biológicos son analizados y comprendidos. No solo amplifican la eficiencia y reproducibilidad en los estudios, sino que también permiten alcanzar objetivos científicos que serían difíciles de lograr mediante enfoques más manuales. Sin embargo, es crucial que los investigadores se familiaricen con las herramientas disponibles y seleccionen cuidadosamente aquellas que se ajusten a sus necesidades analíticas específicas.
Un pipeline en bioinformática es una herramienta indispensable en la era de la genómica y el análisis masivo de datos. Al facilitar procesos complejos y automatizar tareas repetitivas, los pipelines permiten que los investigadores se concentren más en la interpretación de los datos y en la generación de hipótesis significativas. A medida que la tecnología avanza y las capacidades de procesamiento aumentan, la importancia de los pipelines en el ecosistema bioinformático solo seguirá creciendo.
Si quieres conocer otros artículos parecidos a Definición y Función de un Pipeline en Bioinformática puedes visitar la categoría Software.




Deja una respuesta