Estrategias de bioinformática para analizar datos de RNA-Seq
El campo de la bioinformática ha revolucionado la manera en que entendemos y analizamos los datos biológicos, y uno de sus métodos más innovadores es el análisis de datos de RNA-Seq. Esta técnica permite a los investigadores estudiar la expresión génica a un nivel de detalle sin precedentes, proporcionando información valiosa sobre la función celular y los mecanismos de enfermedades. A medida que la tecnología avanza, la cantidad de datos generados por RNA-Seq crece exponencialmente, lo que hace que la aplicación de estrategias bioinformáticas sea esencial para extraer significados biológicos significativos de estos datos complejos.
En este artículo, exploraremos en profundidad las diferentes estrategias de bioinformática empleadas para el análisis de datos de RNA-Seq. Discutiremos desde los pasos fundamentales de la preparación de muestras hasta el análisis y la interpretación de los resultados, así como las herramientas y algoritmos más utilizados en cada fase del proceso. Además, abordaremos los desafíos que se presentan y cómo superarlos, brindando una visión clara y detallada para aquellos que se adentran en este apasionante campo de la biología molecular.
Preparación y calidad de las muestras para RNA-Seq
El éxito de cualquier análisis de RNA-Seq comienza con la obtención de muestras de alta calidad. La recolección y procesamiento adecuado de muestras biológicas es crucial, ya que cualquier error en esta etapa podría afectar drásticamente los resultados del análisis. Comenzamos con la elección del tipo de muestra; las muestras pueden ser de tejido, células o fluidos biológicos, y cada una de ellas presenta sus propios desafíos. Es fundamental que las muestras sean conservadas y procesadas correctamente, para evitar la degradación del RNA.
Una vez obtenidas las muestras, se realiza la extracción de RNA. Este proceso debe llevarse a cabo en condiciones controladas para minimizar la degradación del RNA. Utilizar kits de extracción de alta calidad y asegurarse de que se sigan estrictamente las instrucciones del fabricante es esencial para conseguir un RNA limpio y libre de contaminantes. Después de la extracción, es crucial evaluar la calidad del RNA utilizando métodos como el análisis en gel o la espectroscopia. La integridad del RNA puede determinar si se pueden obtener datos precisos en el análisis posterior.
Secuenciación y generación de datos de RNA-Seq
Una vez que se cuenta con muestras de RNA de alta calidad, el siguiente paso es la secuenciación. Esta etapa implica convertir los fragmentos de RNA en bibliotecas de secuencias que luego se pueden leer mediante plataformas de secuenciación de alto rendimiento, como Illumina o Ion Torrent. La elección de la plataforma de secuenciación puede influir en la calidad y cantidad de datos obtenidos. Por ejemplo, algunas plataformas pueden producir lecturas más largas, lo que facilita la identificación de isoformas de RNA y variantes, mientras que otras pueden proporcionar una mayor profundidad de cobertura.
Es clave durante la generación de datos tener en cuenta los parámetros de calidad establecidos, como la longitud de las lecturas y la profundidad de la secuenciación, para garantizar que se obtenga una representación precisa de la expresión génica. Los datos obtenidos por RNA-Seq son enormes y complejos, lo que hace que el uso de software especializado, compatible con el análisis de datos masivos, sea indispensable en este punto. Los formatos de datos, como FASTQ, deben ser gestionados adecuadamente para poder realizar el análisis posterior.
Procesamiento de datos crudos y alineación
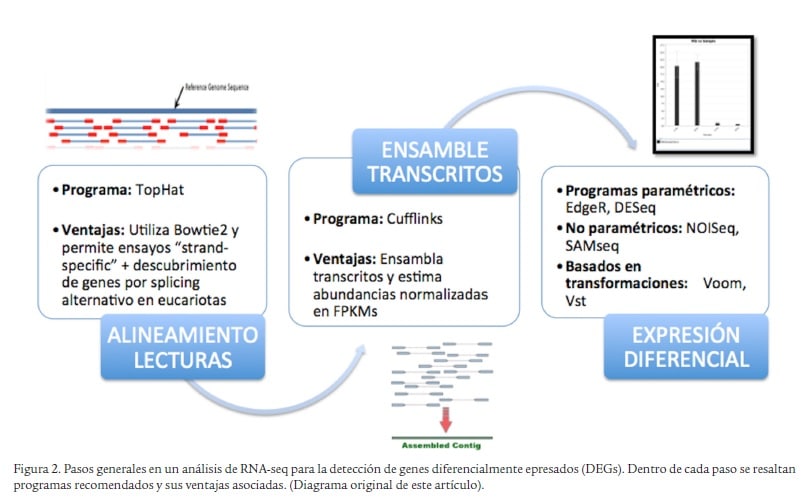
El procesamiento de los datos crudos es una etapa crítica en el análisis de RNA-Seq. Consiste en varios pasos, entre ellos la limpieza de datos, la eliminación de secuencias de baja calidad y la eliminación de adaptadores. Estas tareas son esenciales para asegurar que las lecturas que se utilizan en el análisis sean de alta calidad y representativas del RNA extraído. Herramientas como FastQC y Trimmomatic son comúnmente utilizadas para realizar estas tareas de calidad de datos. Una vez que los datos han sido limpiados, el siguiente paso es la alineación de las lecturas contra un genoma de referencia o un transcriptoma.
La alineación se lleva a cabo utilizando herramientas como STAR o HISAT2, que son altamente eficientes en el manejo de lecturas largas e intrones en genes. El resultado de esta alineación es un archivo que proporciona información sobre dónde encajan las lecturas en el genoma, lo que permite el cálculo preciso de la expresión génica. Sin embargo, es importante abordar el problema de los múltiples alineamientos, donde una lectura puede coincidir con diferentes lugares en el genoma. Algunas estrategias incluyen la elección de una única ubicación o la asignación basada en la proporción de lecturas que van a cada ubicación.
Cuantificación de la expresión génica
Una vez que se han alineado las lecturas, la siguiente fase es la cuantificación de la expresión génica. Esto implica contar cuántas lecturas se alinean con cada gen o transcrito en el transcriptoma. Herramientas como featureCounts o HTSeq son comúnmente utilizadas en esta fase para realizar la cuantificación. El resultado de este paso es un conjunto de datos que indica la cantidad de cada transcrito en la muestra analizada, lo que proporciona una visión clara de la actividad génica bajo condiciones experimentales específicas.
La cuantificación no solo permite determinar qué genes están activos, sino también cuán activos están, lo cual es esencial para entender la respuesta celular a situaciones específicas. Además, esta información se puede utilizar en comparación entre diferentes muestras para identificar cambios en la expresión génica asociados con tratamientos, condiciones ambientales o patologías. Sin embargo, es importante tener en cuenta la normalización de datos, que ayuda a reducir la variabilidad técnica y garantizar que los resultados sean comparables.
Analisis estadístico y biológico de los datos
El análisis estadístico es una parte fundamental del análisis de datos de RNA-Seq. Una vez que se obtiene la cuantificación de la expresión génica, es necesario aplicar métodos estadísticos para identificar genes que están significativamente regulados. Herramientas como DESeq2 y edgeR son comúnmente empleadas para este propósito. Estas herramientas utilizan modelos estadísticos para calcular la variabilidad en los datos y determinar los cambios en la expresión génica que son estadísticamente significativos entre grupos de tratamiento o condiciones experimentales.
El análisis biológico, por otro lado, permite la interpretación de los datos en términos del contexto biológico. Esto puede incluir la realización de análisis de enriquecimiento de rutas metabólicas, donde se evalúa si ciertos grupos de genes se están expresando diferencialmente en función de sus funciones biológicas. Herramientas como GSEA (Gene Set Enrichment Analysis) o DAVID son ampliamente utilizadas para este tipo de análisis. A través de estos enfoques, los investigadores pueden identificar no solo los genes implicados, sino también las vías biológicas que podrían estar bajo regulación en las muestras analizadas.
Desafíos y consideraciones en el análisis de RNA-Seq
A pesar de las potentes herramientas y técnicas disponibles, el análisis de datos de RNA-Seq presenta diversos desafíos. Uno de los obstáculos más significativos es la alta variabilidad biológica y técnica que puede influir en los datos. Es crucial tener un diseño experimental riguroso que considere la replicación y los controles adecuados para poder hacer interpretaciones válidas de los resultados.
Otro aspecto a considerar es la diversidad de transcriptos y sus isoformas presentes en las muestras. Los algoritmos de alineación y cuantificación deben ser lo suficientemente robustos para manejar esta complejidad. Además, la interpretación biológica de los datos puede ser desafiante, ya que identificar la función exacta de un gen en un contexto biológico más amplio requiere un marco de referencia que a menudo no está disponible. Las plataformas y recursos biológicos son esenciales para ayudar en esta interpretación, así como la validación experimental para confirmar hallazgos significativos obtenidos por RNA-Seq.
Conclusiones sobre RNA-Seq y su análisis bioinformático
El análisis de datos de RNA-Seq es una herramienta poderosa que permite a los investigadores desentrañar la complejidad de la expresión génica con gran detalle. Desde la preparación de muestras hasta la interpretación de resultados, cada paso requiere atención cuidadosa y una comprensión profunda de las estrategias bioinformáticas disponibles. A medida que la tecnología avanza y las capacidades de secuenciación mejoran, el uso de estas estrategias seguirá siendo esencial para aprovechar al máximo la información contenida en los datos de RNA-Seq.
Al dominar las técnicas adecuadas y entender los desafíos que surgen en este proceso, los investigadores estarán mejor equipados para contribuir al creciente cuerpo de conocimiento en el campo de la genética y la biología molecular. Así, el continuo desarrollo en métodos bioinformáticos seguirá impulsando los descubrimientos científicos y, en última instancia, mejorando nuestra comprensión de diversas condiciones biológicas y enfermedades.
Si quieres conocer otros artículos parecidos a Estrategias de bioinformática para analizar datos de RNA-Seq puedes visitar la categoría Aplicaciones.

Deja una respuesta