Guía completa sobre el software de ensamblaje de genomas
El **software de ensamblaje de genomas** ha revolucionado la biología moderna y la investigación genética, facilitando un acercamiento sin precedentes a la comprensión de la genética y el funcionamiento de los organismos. A medida que la tecnología avanza, los científicos pueden decodificar los genomas de diferentes especies, en ocasiones en tiempo real, lo que abre nuevas puertas a la investigación en diversas áreas, como la medicina, la agricultura y la conservación de especies. Con este poder también vienen desafíos significativos en términos de procesamiento de datos y precisión, lo que hace que elegir el software adecuado sea crucial para la comunidad científica.
En este artículo, exploraremos en profundidad el **software de ensamblaje de genomas**, sus diversas aplicaciones, las tecnologías involucradas, los desafíos que enfrenta y cómo se está integrando en el panorama de la investigación científica actual. A medida que profundizamos en cada uno de estos aspectos, también consideraremos los factores que los investigadores deben tener en cuenta al seleccionar una herramienta adecuada para sus necesidades específicas. Sin duda, este tema no solo es importante para los genetistas, sino que también tiene implicaciones significativas para la salud pública, la agricultura sostenible y la conservación del medio ambiente.
¿Qué es el ensamblaje de genomas?
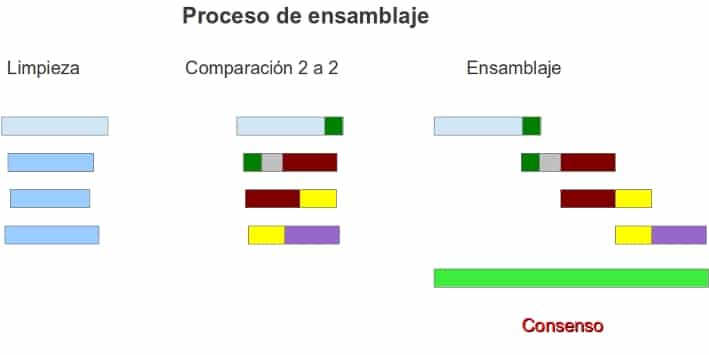
El **ensamblaje de genomas** es el proceso de reconstrucción de la secuencia de un genoma a partir de datos de secuenciación. Este procedimiento involucra la combinación de fragmentos cortos de ADN que se obtienen mediante secuenciación para crear una representación más larga y continua del genoma original. Utilizando algoritmos avanzados y potentes computadoras, se pueden unir estos fragmentos en una secuencia completa. Hay dos principales enfoques de ensamblaje: el **ensamblaje de novo**, que se utiliza cuando no se tiene un genoma de referencia, y el **ensamblaje basado en referencia**, que se usa cuando se tiene acceso a un genoma previamente secuenciado.
El ensamblaje de novo permite a los investigadores construir genomas de organismos poco estudiados, mientras que el ensamblaje basado en referencia es útil para organismos semejantes. La elección entre estos dos metodologías depende de la naturaleza de la investigación, la disponibilidad de recursos y los objetivos específicos del estudio. En la actualidad, existen múltiples herramientas de software que se utilizan para el ensamblaje de genomas, cada una con sus propias características y ventajas.
Tecnologías de secuenciación
La eficacia del **software de ensamblaje de genomas** está intrínsecamente ligada a las tecnologías de secuenciación de ADN que se utilizan. Entre las más prominentes se encuentran la secuenciación de **Sanger**, la secuenciación de **nueva generación (NGS)** y la **sec uenciación de tercera generación (TGS)**. Cada una de estas tecnologías tiene su propio conjunto de ventajas y desventajas que pueden influir en la calidad del ensamblaje resultante.
La secuenciación de Sanger, que ha sido la norma durante décadas, proporciona datos de alta precisión, aunque a un costo elevado y con una capacidad de secuenciación limitada. En contraste, las tecnologías de NGS han permitido secuenciar millones de fragmentos cortos de ADN simultáneamente a un costo mucho más bajo, pero con un mayor margen de error en la fase de ensamblaje. Por último, la secuenciación de tercera generación ofrece lecturas más largas, lo que puede superar algunos de los problemas de ambigüedad encontrados en los enfoques de lectura corta.
Por lo tanto, la elección de la tecnología de secuenciación adecuada no solo afecta directamente el **ensamblaje del genoma**, sino que también determina el tipo de software que se debe utilizar para obtener los mejores resultados. Cada plataforma tiene sus requisitos específicos en términos de pre-procesamiento y ensamblaje, algo que los investigadores deben considerar cuidadosamente.
Principales software de ensamblaje de genomas
En el vasto campo del software de ensamblaje de genomas, hay varias herramientas prominentes que los investigadores utilizan comúnmente. Algunas de las más conocidas incluyen **SPAdes**, **SOAPdenovo**, **Canu** y **Trinity**. Cada una de estas herramientas tiene sus fortalezas únicas y casos de uso específicos.
**SPAdes** es conocido por su capacidad para ensamblar datos de secuenciación de NGS de diversos orígenes, incluyendo datos de **lectura corta** y **lectura larga**. Su diseño modular y su enfoque de ensamblaje por pasos lo hacen ideal para proyectos grandes y complejos. Por su parte, **SOAPdenovo** también es popular en proyectos de ensamblaje de novo, conocido por su rapidez y eficacia, aunque puede requerir una configuración más compleja.
**Canu** es otra importante herramienta destacada principalmente en el ensamblaje de secuencias de **lectura larga**, haciéndola útil para el ensamblaje de genomas de organismos más grandes y complejos. En los proyectos de |* transcriptómica**, **Trinity** ha demostrado ser una opción robusta y eficaz, agrupando las lecturas en **transcritos** y facilitando el análisis funcional y la anotación.
La elección y uso de estas herramientas dependerán, en gran medida, de las necesidades específicas de cada proyecto, así como de los conocimientos técnicos del equipo de investigación. Es crucial realizar un análisis detallado de las métricas de rendimiento y la calidad de datos que brinda cada software.
Desafíos en el ensamblaje de genomas
A pesar de los avances en el **ensamblaje de genomas**, todavía existen múltiples desafíos que los investigadores deben enfrentar. Uno de los más prominentes es la **complejidad del genoma**. Genomas de organismos con estructuras complejas, grandes cantidades de repetición o poliploides pueden resultar en dificultades durante el ensamblaje, ya que los métodos existentes pueden no ser lo suficientemente efectivos.
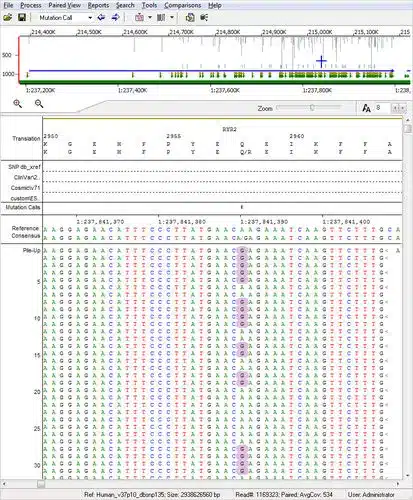
Además, los **errores de secuenciación** son un problema inherente en todas las tecnologías de secuenciación. Estos errores pueden llevar a la creación de ensamblajes equivocados, lo que a su vez impide un análisis y una interpretación precisos. La calidad de los datos originales es fundamental, lo que lleva a un ciclo continuo de mejora en las metodologías de secuenciación y ensamblaje.
Finalmente, la interpretación de los datos del ensamblaje es una tarea que requiere un alto nivel de conocimiento especializado. Los investigadores no solo deben dominar el software, sino que también deben contar con la comprensión necesaria de las herramientas bioinformáticas, la biología molecular y la genética para hacer conclusiones válidas y efectivas a partir de los genomas ensamblados.
El futuro del ensamblaje de genomas
El futuro del **ensamblaje de genomas** y su software promete ser aún más emocionante. La integración de tecnologías avanzadas como la inteligencia artificial y el aprendizaje automático está comenzando a transformar la forma en que se manejan y analizan los datos de secuenciación. Estas tecnologías pueden ayudar a mejorar la exactitud del ensamblaje y a reducir el impacto de los errores de secuenciación, optimizando el proceso de reconstrucción de genomas.
Además, la explosión de datos de secuenciación generados a través de iniciativas como el Proyecto del Genoma Humano 100 000 Genomas y la creciente accesibilidad de tecnologías de secuenciación de vanguardia contribuirán a un mejor ensamblaje de genomas complejos. También se espera que el desarrollo de software sea más intuitivo, permitiendo a los investigadores acceder y usar eficientemente estas herramientas sin necesidad de profundos conocimientos en bioinformática.
Conclusión
El **software de ensamblaje de genomas** es una parte integral de la biología moderna y la investigación genética, desempeñando un papel crucial en la interpretación de la secuencia de ADN y en la comprensión de los organismos que habitan nuestro planeta. A medida que la ciencia avanza, las tecnologías de secuenciación continúan evolucionando, y con ello, las herramientas y técnicas que los científicos utilizan para ensamblar genomas de manera efectiva.
A través de este artículo, hemos explorado las complejidades del ensamblaje de genomas, desde los enfoques tecnológicos y el software más utilizado, hasta los desafíos y el futuro del campo. La constante innovación en este ámbito promete un futuro brilloso, donde los descubrimientos sobre genética y biología serán más accesibles y comprensibles que nunca. Sin duda, el camino del ensamblaje de genomas está lleno de posibilidades emocionantes y trascendentales que beneficiarán a la salud, la agricultura y la conservación del medio ambiente a largo plazo.
Si quieres conocer otros artículos parecidos a Guía completa sobre el software de ensamblaje de genomas puedes visitar la categoría Software.




Deja una respuesta