Redes de co-regulación en análisis de transcriptómica: qué son
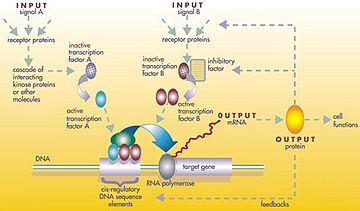
Las redes de co-regulación en análisis de transcriptómica son una herramienta fundamental en la biología molecular que permite comprender cómo las diferentes moléculas de ARN interactúan y se regulan entre sí. La transcriptómica, que estudia el conjunto de ARN presentes en una célula, ha dado un giro revolucionario a nuestra comprensión del funcionamiento celular, y las redes de co-regulación ofrecen un marco para analizar las interacciones complejas que ocurren en el interior de las células. En un mundo donde los datos genómicos se multiplican, la capacidad de analizar y conectar esta información se vuelve crucial para avanzar en la investigación biomédica y el desarrollo de nuevas terapias.
En este artículo, nos adentraremos en el fascinante mundo de las redes de co-regulación en el análisis de transcriptómica. Abordaremos qué son, cómo se construyen y cuáles son sus aplicaciones en la investigación biomédica. Además, profundizaremos en las técnicas y herramientas utilizadas para construir estas redes, así como en los desafíos y oportunidades que presentan en la actualidad. Al final, se espera que el lector comprenda la importancia de estas redes en la biología moderna y su impacto en la investigación, así como los posibles caminos futuros que se pueden explorar a través de esta apasionante área.
Qué son las redes de co-regulación
Las redes de co-regulación son representaciones gráficas que ilustran cómo los diferentes genes y sus productos interactúan entre sí y se regulan mutuamente en un sistema biológico. Estas redes se basan en la premisa de que la expresión de un gen puede estar influenciada por múltiples factores, incluyendo otros genes, proteínas y siRNA. La co-regulación se puede observar en diversas formas, como la regulación en cascada, en la que un gen activa o reprime la expresión de otro, o en regulaciones en red, donde varios genes actúan conjuntamente para controlar un fenotipo específico.
Las investigaciones han demostrado que estas redes no son estructuralmente aleatorias, sino que poseen una arquitectura organizada que puede reflejar funciones biológicas específicas. Además, estas redes son dinámicas, lo que significa que pueden cambiar en respuesta a diferentes estímulos, como el estrés o la diferenciación celular. Por tanto, estudiar las redes de co-regulación permite entender no solo cómo se regulan los genes, sino también cómo esto afecta la función celular en diversos contextos fisiológicos y patológicos.
Construcción de redes de co-regulación
La construcción de redes de co-regulación es un proceso complejo que implica la recopilación y análisis de datos transcriptómicos. Estos datos se obtienen a través de diferentes tecnologías de secuenciación de ARN que permiten medir la cantidad de ARN mensajero (ARNm) presente en una célula en diversas condiciones. Una vez se han obtenido los datos, se aplican diversas técnicas de análisis bioinformático, como la correlación de expresión, para identificar la co-regulación entre genes.
La correlación de expresión es una técnica estadística que permite evaluar el grado en el que dos genes muestran patrones de expresión similares bajo diferentes condiciones. Esto se puede llevar a cabo utilizando métodos de correlación como el coeficiente de correlación de Pearson o de Spearman. La interpretación de estos resultados es crucial, ya que una alta correalción no siempre indica un mecanismo de regulación directo, sino que podría ser resultado de interacciones indirectas o influencias comunes en el ambiente de la célula.
Una vez que se han recopilado suficientes datos de correlación, se puede proceder a la construcción de la red utilizando software especializado, como Cytoscape o Gephi. Estas herramientas permiten visualizar las interacciones entre genes y explorar la estructura global de la red. Es importante señalar que la validación experimental de las predicciones obtenidas a través de modelos computacionales es un paso vital para confirmar la existencia de las interacciones propuestas.
Aplicaciones de redes de co-regulación en investigación biomédica
Las redes de co-regulación tienen una amplia variedad de aplicaciones en el campo de la investigación biomédica. Una de las áreas más prometedoras es la medicina personalizada, donde la comprensión de las redes de expresión de genes puede ayudar a predecir cómo un paciente individual responderá a un tratamiento. Al identificar las rutas de regulación y los genes involucrados en la respuesta a terapias específicas, los investigadores pueden desarrollar enfoques más dirigidos y eficaces en el tratamiento de enfermedades, especialmente en el ámbito del cáncer.
Otras aplicaciones incluyen la identificación de biomarcadores para enfermedades, donde la alteración en patrones de expresión de ciertos genes dentro de la red puede indicar la presencia o progresión de una enfermedad. También se utilizan las redes de co-regulación para estudiar mecanismos de resistencia a fármacos, permitiendo a los científicos averiguar cómo ciertas células pueden evadir la muerte inducida por tratamiento, lo cual es crucial para el desarrollo de estrategias que superen esta resistencia.
El estudio de redes de co-regulación también ha contribuido significativamente al entendimiento de enfermedades complejas, como enfermedades neurodegenerativas y trastornos metabólicos. Comprender cómo se interrelacionan los diferentes genes en estas patologías puede proporcionar información valiosa sobre su fisiopatología y potencialmente abrir caminos hacia nuevas opciones terapéuticas.
Desafíos en el análisis de redes de co-regulación
A pesar de sus numerosas ventajas, el análisis de redes de co-regulación presenta varios desafíos. Uno de los principales problemas es la interpretación de los datos, ya que la dirección y la naturaleza de las interacciones a menudo son difíciles de determinar. Esencialmente, la correlación no implica causalidad, y se requiere un enfoque multifacético para agotar todas las posibilidades. Además, la variabilidad biológica entre diferentes muestras y condiciones experimentales puede complicar aún más la interpretación de los resultados.
Otro desafío es el manejo inmenso de datos implicado en el análisis de redes. Con el avance de las tecnologías de secuenciación, la cantidad de datos que se generan es monumental, y se requiere un hardware y software potente para procesarlos adecuadamente. La integración de datos de diferentes fuentes, como la genómica, la proteómica y la metabolómica, también representa un reto, pero es vital para crear un panorama más completo del sistema biológico en estudio.
Finalmente, la validación experimental sigue siendo un paso crucial que requiere mucho tiempo y recursos. Los investigadores deben ser diligentes en confirmar la existencia de las interacciones predichas mediante experimentos de laboratorio que pueden ser costosos y laboriosos, lo que puede limitar la velocidad a la que se pueden probar nuevas hipótesis emergentes de los análisis computacionales.
El futuro de las redes de co-regulación
El futuro de las redes de co-regulación es prometedor y emocionante. Con los avances en tecnología de secuenciación y análisis de datos, se espera que se desarrollen nuevas herramientas que permitan a los investigadores construir redes más precisas y completas. La inteligencia artificial y el aprendizaje automático también están comenzando a jugar un papel importante en el análisis de datos biológicos, lo que podría revolucionar la forma en que entendemos la co-regulación genética.
Además, el enfoque en la biología de sistemas, en el que se estudian las interacciones como parte de un todo, podría dar lugar a una comprensión más integral de las redes de co-regulación. Este enfoque tiene el potencial de conectar datos a través de diferentes niveles de organización biológica, desde moléculas individuales hasta la fisiología del organismo y la interacción con el medio ambiente.
Finalmente, a medida que ampliemos nuestro conocimiento sobre las redes de co-regulación, es probable que surjan nuevas aplicaciones en campos que aún no hemos explorado completamente, desde la biotecnología hasta la ecología, donde estas redes también pueden desempeñar un papel crucial. El futuro en este campo promete enriquecer nuestra comprensión de la biología y transformar el abordaje de enfermedades complejas y la medicina personalizada.
Conclusión
Las redes de co-regulación en análisis de transcriptómica representan un área fascinante y dinámica de la investigación biológica. Al comprender cómo los genes se regulan entre sí y cómo estas interacciones impactan la función celular, los científicos están abriendo nuevas puertas a la medicina personalizada, la identificación de biomarcadores y el descubrimiento de nuevas terapias. A medida que avanzamos en la recopilación y análisis de datos, enfrentamos desafíos significativos, desde la interpretación de interacciones hasta la validación experimental. Sin embargo, el futuro de la investigación en redes de co-regulación es prometedor y podría proporcionar insights valiosos en diversas áreas de la biología y la medicina. Las redes de co-regulación no solo son una herramienta poderosa en la investigación; son un componente esencial para desentrañar los complejos mecanismos que subyacen en la vida misma.
Si quieres conocer otros artículos parecidos a Redes de co-regulación en análisis de transcriptómica: qué son puedes visitar la categoría Transcriptómica.

Deja una respuesta