Tipos de algoritmos utilizados en análisis filogenético
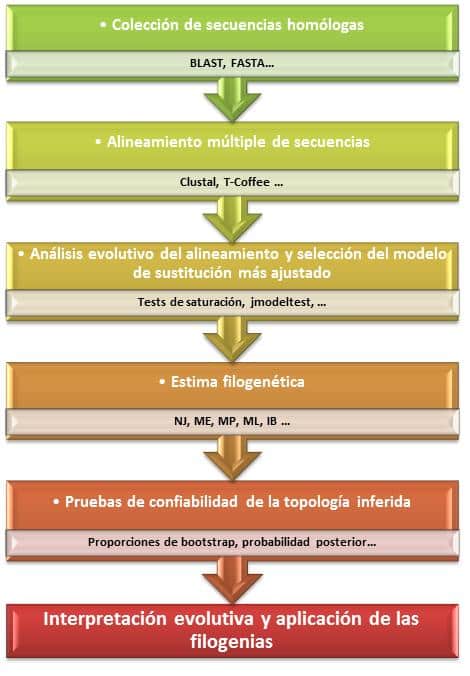
El análisis filogenético es una herramienta fundamental en la biología evolutiva que permite a los científicos interpretar las relaciones entre diferentes especies o grupos de organismos a través del tiempo. Esta disciplina revela no solo las similitudes y diferencias morfológicas y genéticas, sino que también ayuda a entender cómo han evolucionado las especies en el contexto de la historia natural. Examinar los tipos de algoritmos utilizados en análisis filogenético se convierte así en un paso esencial para comprender las metodologías que impulsan esta ciencia, ya que los algoritmos son las bases que permiten construir árboles filogenéticos y estimar relaciones evolutivas.
Este artículo se adentrará en los diversos tipos de algoritmos que se aplican en el análisis filogenético, explorando sus principios, funciones y aplicaciones. Vamos a desglosar cada uno de estos algoritmos y su relevancia en la construcción de árboles filogenéticos, así como sus especificaciones y los contextos en los que se utilizan. A medida que progresamos, evidenciamos cómo cada herramienta contribuye a un entendimiento más profundo sobre la biodiversidad y la historia evolutiva de los organismos.
Algoritmos basados en la distancia
Los algoritmos basados en la distancia son algunos de los más comunes en el análisis filogenético. Estos algoritmos funcionan calculando una medida de distancia entre las secuencias de ADN, proteínas o morfologías de distintos organismos. La distancia, que puede ser euclidiana, de Hamming o Jukes-Cantor, sirve como parámetro para construir un árbol filogenético. El enfoque con este tipo de algoritmos es que las especies o grupos genéticamente similares tienen menores distancias entre ellos, mientras que aquellas que son más divergentes tendrán mayores distancias.
Uno de los algoritmos más representativos de esta categoría es el método de Neighbor-Joining. Este algoritmo crea un árbol filogenético a partir de una matriz de distancias calculadas, agrupando a las especies más cercanas primero, lo que lleva a la formación del árbol a partir de ramas. A través de este proceso, se pueden encontrar los grupos monofiléticos y parafiléticos, permitiendo explorar la divergencia de especies a lo largo del tiempo.
Un aspecto importante a destacar en los algoritmos basados en la distancia es que son generalmente rápidos y eficientes en comparación con otros métodos. Sin embargo, su precisión puede disminuir significativamente si las distancias no reflejan adecuadamente las relaciones evolutivas reales, especialmente en el caso de eventos complejos de evolución genética, como el equilibrio puntuado o la homoplasia.
Algoritmos basados en máximos de verosimilitud
El enfoque de los algoritmos basados en máximos de verosimilitud es más sofisticado y se tiene en armonía con modelos evolutivos más completos, lo que permite evaluar la mejor explicación posible para un conjunto de datos filogenéticos. Este tipo de algoritmo asume un modelo de evolución de las características que puede incluir tasas de cambio, raíces evolutivas y otros parámetros. Esto confiere al análisis una adaptación mucho más precisa a las realidades evolutivas, así como una panorámica más clara de la relación entre las especies.
Uno de los algoritmos más conocidos en esta categoría es el RAxML (Randomized Axelerated Maximum Likelihood), que se ha vuelto esencial en estudios filogenéticos de gran escala. Su capacidad para manejar grandes volúmenes de datos y su eficiencia en el cálculo de modelos hacen que sea una herramienta favorita entre los investigadores. Su uso se extiende a una variedad de políticas evolutivas, permitiendo así que se analicen no solo los datos de ADN, sino también de proteínas o rasgos morfológicos.
Es crucial mencionar que, aunque los algoritmos basados en máximos de verosimilitud son poderosos, requieren de un mayor tiempo de cómputo y son más complejos en su implementación en comparación con los métodos basados en la distancia. Sin embargo, su capacidad para adaptarse a distintos modelos evolutivos y proporcionar resultados altamente consistentes los hace invaluables en el campo del análisis filogenético.
Algoritmos de programaciones bayesianas
El uso de algoritmos de programaciones bayesianas en filogenética ha cobrado fuerza en los últimos años. Estos algoritmos permiten a los investigadores incorporar información previa sobre la evolución de las especies, actualizándola a medida que se adquiere nueva evidencia. Este enfoque probabilístico ayuda a abordar la incertidumbre en los datos y permite generar distribuciones de probabilidad sobre la estructura del árbol filogenético, considerando múltiples escenarios evolutivos.
Uno de los software más valorados en este contexto es MrBayes, que utiliza muestreo de Monte Carlo para calcular la probabilidad posterior de un árbol filogenético dado un conjunto de datos. Su implementación es estándar en muchos campos de la biología, ya que permite explorar las zonas de incerteza y evaluar la confiabilidad de las filogenias generadas. Este enfoque contribuye en la construcción de árboles con métricas como el valor posterior, o credibilidad, que representa la probabilidad de que un grupo de especies esté relacionado dada la información disponible.
A pesar de su complejidad y los requerimientos computacionales que implican, los algoritmos de programación bayesiana ofrecen una forma robusta de manejar la variabilidad y la complejidad inherente a los datos filogenéticos, y su implementación se vuelve esencial en estudios de alta resolución donde cada detalle cuenta.
Comparación de algoritmos en análisis filogenético
Realizar una comparación entre los distintos algoritmos utilizados en análisis filogenético permite entender los beneficios y limitaciones asociados a cada uno. Un método basado en distancia como el Neighbor-Joining es preferido por su rapidez y simplicidad, mientras que los métodos de máxima verosimilitud brindan un ajuste más refinado a los modelos evolutivos y son ideales para conjuntos de datos complejos. Por su parte, los algoritmos bayesianos se destacan por su capacidad para manejar la incertidumbre, siendo cada uno una herramienta imprescindible dependiendo del contexto del análisis.
La elección del algoritmo adecuado depende de varios factores, incluyendo el tamaño del conjunto de datos, la naturaleza de las características que se estudian y el objetivo del análisis. Por ejemplo, para estudios preliminares donde se desea comprender rápidamente las relaciones entre un grupo de especies, un enfoque de distancia puede ser suficiente. Sin embargo, para estudios más detallados donde se requiera una representación precisa de la historia evolutiva, los algoritmos de máxima verosimilitud o bayesianos son altamente recomendables.
Desafíos en el análisis filogenético
A pesar de los avances en los algoritmos utilizados en el análisis filogenético, persisten diversos desafíos. Uno de los principales problemas es la aparición de datos faltantes o ruido en los datos. Estos aspectos pueden distorsionar las inferencias filogenéticas y llevar a conclusiones erróneas sobre las relaciones evolutivas. Además, la homoplasia, el fenómeno donde evolutivamente diferentes especies desarrollan características similares por adaptaciones no relacionadas, complica la interpretación de las filogenias.
De igual forma, los fenómenos de evolución convergente o paralela pueden crear patrones engañosos en los árboles filogenéticos. Por estos motivos, es fundamental combinar diversas metodologías y automatizar rigurosamente el proceso de evaluación de datos para obtener resultados confiables.
Perspectivas futuras en el análisis filogenético
El futuro del análisis filogenético está en la integración de este campo con nuevas tecnologías como el bioinformática y la secuenciación de ADN de alto rendimiento. A medida que avanzamos hacia una era de datos masivos, la capacidad para analizar cientos de miles de secuencias de manera simultánea revolucionará la forma en que interpretamos la historia evolutiva de los organismos. Esto no solo hará que los algoritmos existentes sean aún más relevantes, sino que también abrirá la puerta a nuevos enfoques y metodologías que todavía están en gestación.
También hay un creciente interés por implementar técnicas de inteligencia artificial y aprendizaje automático para mejorar la precisión y eficiencia en la construcción de árboles filogenéticos. Estos enfoques innovadores podrían optimizar cómo se procesan los datos y cómo se interpretan las relaciones evolutivas, lo que traería consigo avances significativos y abriría nuevas posibilidades en investigación biológica.
Conclusión
Los distintos tipos de algoritmos utilizados en análisis filogenético juegan un papel crucial en nuestra comprensión de la historia de la vida en la Tierra. Desde los métodos basados en la distancia hasta los algoritmos de máxima verosimilitud y las programaciones bayesianas, cada enfoque presenta sus ventajas y desventajas, lo que hace que la elección del método adecuado dependa en gran medida del contexto del estudio y del tipo de datos disponibles. Aunque existen desafíos, los avances continuos en la tecnología y los métodos analíticos prometen profundizar nuestra comprensión de la diversidad biológica y las complejidades de la evolución. La evolución de los algoritmos en este campo no solo refleja la constante búsqueda de precisión en la biología, sino que también subraya la importancia de la interdisciplinariedad en la ciencia moderna.
Si quieres conocer otros artículos parecidos a Tipos de algoritmos utilizados en análisis filogenético puedes visitar la categoría Algoritmos.

Deja una respuesta