Validación experimental de SNPs: Métodos e importancia en Genética
La validación experimental de SNPs (polimorfismos de un solo nucleótido) se ha convertido en un pilar fundamental para comprender la variabilidad genética en numerosas especies, incluyendo los humanos. Estos pequeños cambios en la secuencia del ADN pueden tener grandes implicaciones para la salud, la evolución y la adaptación de los organismos. A medida que la ciencia genética evoluciona y avanza en su capacidad para analizar y modelar estas variaciones, la necesidad de validar experimentalmente los SNPs se convierte en una prioridad para investigadores y clínicos por igual.
Este artículo se adentra en los diferentes métodos utilizados para la validación de SNPs y discute su importancia en el campo de la genética. La validación no solo asegura la precisión de los SNPs identificados en estudios genéticos, sino que también fortalece la interpretación de los resultados y sus aplicaciones en diversas áreas, como la medicina de precisión y el mejoramiento genético en plantas y animales. A lo largo de este artículo, examinaremos los enfoques más significativos, así como ejemplos prácticos que resaltan la importancia de esta validación en el contexto de la investigación genética contemporánea.
Comprendiendo los SNPs y su relevancia
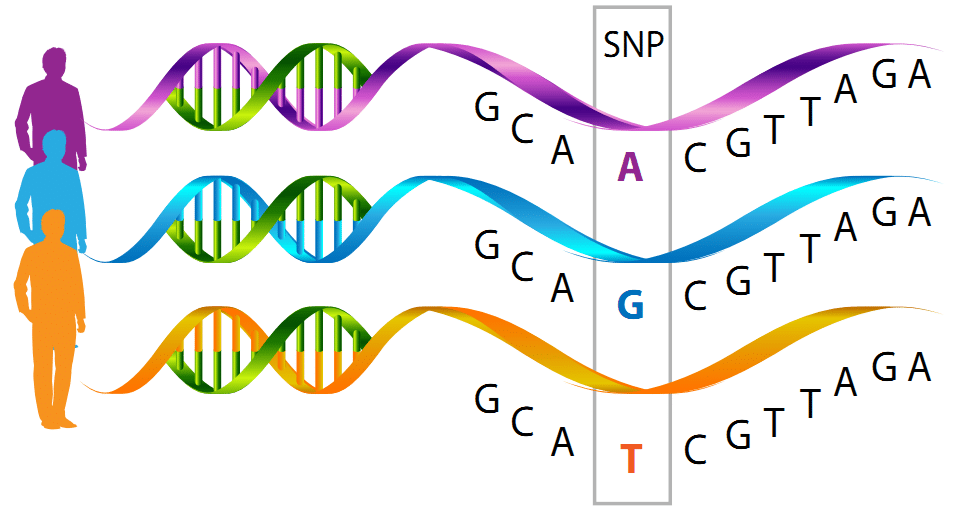
Los SNPs son las variaciones más comunes en el genoma humano y en el de muchas otras especies. Se definen como cambios en una sola base del ADN entre individuos. Por ejemplo, en un lugar específico del genoma, un individuo puede tener una adenina (A) mientras que otro podría tener una guanina (G). Estas variaciones pueden influir en la forma en que se expresa un gen, afectando así características fenotípicas, la predisposición a enfermedades, la respuesta a medicamentos y más. Por lo tanto, entender y validar estos SNPs es crucial para varias aplicaciones, desde la investigación biomédica hasta la agricultura.
Existen miles de millones de SNPs en los genomas humanos, muchos de los cuales han sido identificados a través de tecnologías de secuenciación de última generación. Sin embargo, la presencia de un SNP en una base de datos no garantiza su funcionalidad ni su relevancia biológica. Es aquí donde la validación experimental de SNPs entra en juego, proporcionando la evidencia necesaria para confirmar que un SNP afectará de manera significativa algún rasgo o enfermedad. Esencialmente, la validación permite a los investigadores clasificar y priorizar SNPs para estudios posteriores, asegurando que los más relevantes sean analizados en profundidad.
Métodos de validación experimental de SNPs
La validación experimental de SNPs puede llevarse a cabo utilizando una amplia variedad de técnicas y métodos, cada uno con sus propias ventajas y desventajas. Entre los métodos más comunes están la secuenciación de Sanger, los análisis de genotipado y las pruebas funcionales. A continuación, exploraremos cada uno de estos métodos en detalle.
Secuenciación de Sanger
La secuenciación de Sanger es considerada el estándar de oro para la validación de SNPs. Este método utiliza la terminación de cadenas de ADN para determinar la secuencia exacta de nucleótidos en una muestra. Aunque su costo y tiempo requerido son mayores en comparación con otras técnicas más modernas, su precisión y fiabilidad hacen que sea muy valorado en estudios de validación. La secuenciación de Sanger permite a los investigadores producir una lectura clara del ADN, identificando SNPs con alta certeza y resolviendo ambigüedades presentadas en estudios iniciales.
A través de este método, los SNPs pueden ser validados directamente en las muestras de ADN de individuos seleccionados. La secuenciación de Sanger garantiza que solo los SNPs que son realmente funcionales y relevantes sean seleccionados para su análisis posteriores o para su uso en aplicaciones clínicas. Sin embargo, la desventaja principal es que este método es laborioso y no es escalable para el análisis de grandes muestras, lo que ha llevado a la búsqueda de métodos más eficientes.
Análisis de genotipado
Un método alternativo y mucho más eficiente es el análisis de genotipado, que permite la detección de múltiples SNPs en una sola reacción. Existen varias plataformas para el genotipado, incluida la tecnología de microarreglos y la PCR en tiempo real. Estas técnicas permiten el análisis rápido y en gran escala de SNPs, facilitando el trabajo con grandes cohortes de datos.
No obstante, el genotipado tiene sus propias limitaciones, ya que, aunque puede detectar variantes de manera eficiente, la especificidad de la técnica puede no igualar la precisión que se obtiene a través de la secuenciación de Sanger. Por lo tanto, muchos estudios optan por una combinación de ambas metodologías: utilizando el análisis de genotipado para una detección inicial y luego validando los resultados con secuenciación de Sanger para confirmar los SNPs más relevantes.
Pruebas funcionales
La validación de SNPs no se detiene únicamente en la detección de variantes. Las pruebas funcionales son igualmente vitales para entender el impacto que los SNPs pueden tener en el fenotipo. Estos ensayos se llevan a cabo en sistemas modelo, como células en cultivo o en organismos transgénicos, donde se puede manipular el SNP en cuestión y observar los efectos de la variación en el funcionamiento celular o en el desarrollo del organismo.
Por ejemplo, si un SNP se asocia con un riesgo elevado de enfermedad, realizar pruebas funcionales podría demostrar cómo esa variante particular afecta la expresión del gen o el funcionamiento de la proteína resultante. Este enfoque no solo valida la asociación, sino que también proporciona una comprensión más rica y contextual de los mecanismos biológicos subyacentes. Sin embargo, realizar estas pruebas puede ser costoso y laborioso, lo que a menudo limita su uso a SNPs que ya han demostrado una clara asociación en estudios previos.
Importancia de la validación experimental en el contexto clínico
Conforme la medicina personalizada y la farmacogenómica toman más fuerza, la importancia de la validación experimental de SNPs se vuelve aún más crítica. Las implicaciones para la salud humana son extraordinarias, especialmente cuando se considera el potencial de los SNPs para influenciar la respuesta a tratamientos o el riesgo de desarrollar ciertas enfermedades. Por ejemplo, un SNP que modula la metabolización de un fármaco puede transformar por completo las pautas de tratamiento para una condición médica específica.
La validación asegura que los estudios que identifican asociaciones entre SNPs y fenómenos clínicos sean robustos y replicables. Sin validación, un estudio podría generar falsos positivos, llevando a la adopción de enfoques de tratamiento que no son eficaces, o incluso dañinos. Por lo tanto, la validación experimental no solo es un paso esencial en la investigación genética, sino que también puede ser de vital importancia para la creación de pautas clínicas basadas en la genética.
Conclusión
La validación experimental de SNPs representa una etapa crucial en la investigación genética, asegurando que las variaciones genéticas identificadas sean verdaderamente relevantes y funcionales. A través de métodos como la secuenciación de Sanger, el análisis de genotipado y las pruebas funcionales, los investigadores pueden construir una base sólida para la comprensión de la variabilidad genética y su impacto en la salud y la enfermedad. En un mundo donde la medicina personalizada se está convirtiendo en un objetivo primario, la validación de SNPs se presenta como un elemento indispensable para garantizar que los tratamientos basados en genómica sean usables, seguros y eficaces. A medida que la tecnología y los métodos continúan avanzando, es seguro que el campo de la validación de SNPs seguirá evolucionando, abriendo nuevas vías para descubrimientos e innovaciones en la genética y más allá.
Si quieres conocer otros artículos parecidos a Validación experimental de SNPs: Métodos e importancia en Genética puedes visitar la categoría SNPs.




Deja una respuesta